NVIDIA Clara Parabricks v3 . 6 的發布為其強大的基因組分析工具套件帶來了新的變體調用、注釋、篩選和質量控制應用。現在,在基因組分析的每個階段都有超過 33 個加速工具, NVIDIA Clara Parabricks 提供 GPU 加速的生物信息管道,可以擴展到任何工作負載。
由于基因組和外顯子的測序速度比以往任何時候都快,必須對越來越多的原始儀器數據進行映射、對齊和解釋,以破譯變異及其對疾病的意義。生物信息管道需要跟上基因組分析工具的步伐。基于 CPU 的分析管道通常需要數周或數月的時間收集結果,而基于 GPU 的管道可以在 22 分鐘內分析 30 倍全人類基因組,在 4 分鐘內分析全人類外顯子。
這些快速的周轉時間對于跟上下一代測序( NGS )基因組儀器的輸出是必要的。這對于大規模人口、癌癥中心、 ph ARM 藥物開發和基因組研究項目來說是必不可少的,因為這些項目需要出版物的快速結果。
NVIDIA Clara Parabricks v3 . 6 包含:
- 新 GPU – 加速變體調用者
- 一個易于使用的基于投票的 VCF 合并工具( VBVM )
- 數據庫注釋工具( VCFANNO )
- 通過等位基因頻率快速過濾 VCF 的新工具( FrequencyFilter )
- 用于體細胞和種系管道的 VCF 質量控制工具( VCFQC 和 VCFQCbyBAM )。
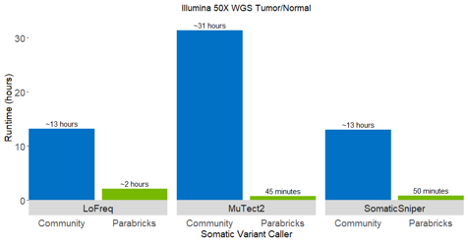
加速 LoFreq 和其他軀體來電者
隨著 LoFreq 與 Strelka2 、 Mutect2 和 SomaticSniper 一起加入, Clara Parabricks 現在包括 4 個癌癥工作流程的軀體細胞呼叫者。 LoFreq 是一個快速而敏感的變量調用程序,用于從 NGS 數據推斷 SNV 和 INDEL 。它可以自動適應覆蓋率和測序質量的變化,并可應用于體細胞、病毒/準種、宏基因組和細菌數據集。
Clara Parabricks 中的 Lofreq 體細胞調用程序比其本地實例快 10 倍,非常適合調用低頻突變。使用基本調用質量和 NGS 數據中固有的其他錯誤源, Lofreq 提高了調用低于 10% 等位基因頻率閾值的體細胞突變的準確性。
在 v3 . 6 中, 經過加速的 LoFreq 僅支持 SNV 調用,而 Indel 調用將在后續版本中提供。
閱讀更多>>
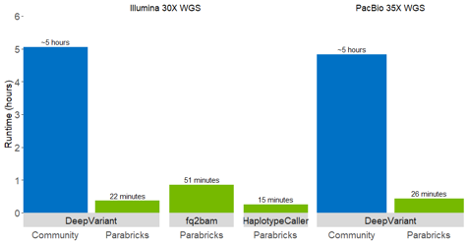
使用新的加速工具從數月到數小時
NVIDIA Clara Parabricks v3 . 6 還包括一個 bam2fastq 工具,添加了 smoove 變體調用程序,支持從頭突變,以及用于 VCF 處理的新工具(例如注釋、篩選和合并)。對 30 倍人類基因組的標準 WGS 分析在DGX A100上完成,耗時 22 分鐘,比同一服務器上基于 CPU 的工作流快 80 多倍。通過這種加速,耗時數月的項目現在可以在數小時內完成。
Bam2Fastq 是 GATK Sam2fastq 的加速版本。它將 BAM 或 CRAM 文件轉換為 FASTQ 。這對于需要將樣本重新對齊到新引用,但刪除原始 FASTQ 以節省存儲空間的場景非常有用。現在,它們可以從 BAM 中重新生成,并比以前更快地與新引用對齊
在比較后代與其父母的序列數據時,檢測生殖系基因組中發生的從頭變異( DNV )(也稱為三重分析)對于疾病相關變異的研究以及建立世代突變率的基線至關重要。
Parabricks Clara Clara v3 . 6 中包含了一個基于 GPU 的調用 DNV 的工作流,該工作流利用了谷歌的 DeepVariant ,它已經在 trio 分析和其他譜系測序項目中進行了測試。
了解更多>>
對于結構變體調用, NVIDIA Clara Parabricks 已經包括 Manta ,現在添加了 smoove 。 Smoove 簡化并加快了短讀結構變體的調用和基因分型。它還通過去除指示低電平噪聲的對準信號來提高特異性,并且常常導致虛假呼叫。
了解更多>>
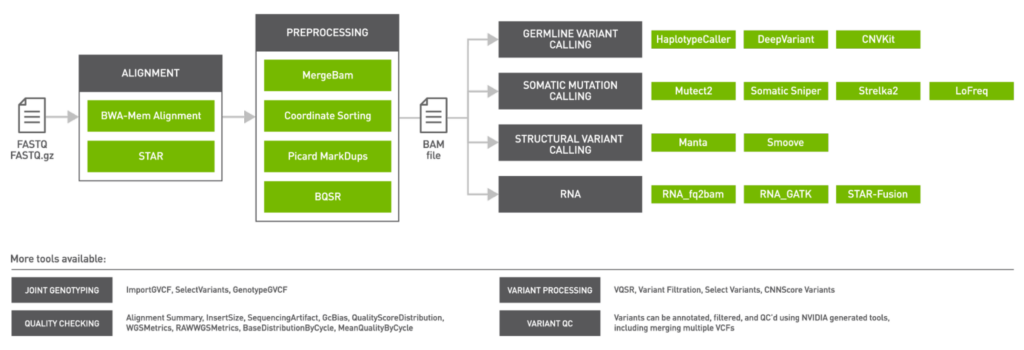
NVIDIA Clara Parabricks v3 . 6 還關注了變異調用后基因組管道的步驟。 BamBasedVCFQC 是一個 NVIDIA 生成的工具,通過使用原始 BAM 的 SamTools mPileUp 結果來幫助 QC VCF 輸出。 Vcfanno 允許用戶使用第三方數據源(如 dbSNP )注釋 VCF 輸出,向 VCF 添加等位基因頻率。
FrequencyFiltering 允許根據包含等位基因頻率和讀取計數信息的數字字段過濾 VCF 中的變量。最后,基于投票的體細胞呼叫者合并( vbvm )用于合并兩個或多個 VCF 文件,然后基于簡單的基于投票的機制過濾變體,其中變體可以基于已識別特定變體的體細胞呼叫者的數量進行過濾。
- 請嘗試 NVIDIA Clara Parabricks GPU – 用于種系、癌癥和 RNA 序列分析工作流的加速基因組分析工具免費使用 90 天試用許可證>>
- 通過AWS 市場>>
- 閱讀 v3 . 6 上的更多信息>
?