不同領域的數據科學家使用聚類方法在他們的數據集中找到自然的“相似”觀察組。流行的聚類方法可以是:
- 基于質心的:根據與某個質心的接近程度將點分組為 k 組。
- 基于圖形的:根據圖中頂點的連接對其進行分組。
- Density-based:根據附近區域數據的密度或稀疏性更靈活地分組。
基于層次密度的應用程序空間聚類 w / Noise (HDBSCAN)算法是一種density-based聚類方法,對噪聲具有魯棒性(將稀疏區域中的點作為簇邊界,并將其中一些點直接標記為噪聲)。基于密度的聚類方法,如 HDBSCAN ,能夠發現形狀奇特、大小各異的聚類 — 與k-means、k-medioids或高斯混合模型等基于質心的聚類方法截然不同,這些方法找到一組 k 個質心,將簇建模為固定形狀和大小的球。除了必須預先指定 k 之外,基于質心的算法的性能和簡單性幫助它們仍然是高維聚類點的最流行方法之一;即使在不修改輸入數據點的情況下,它們也無法對不同大小、形狀或密度的簇進行建模。
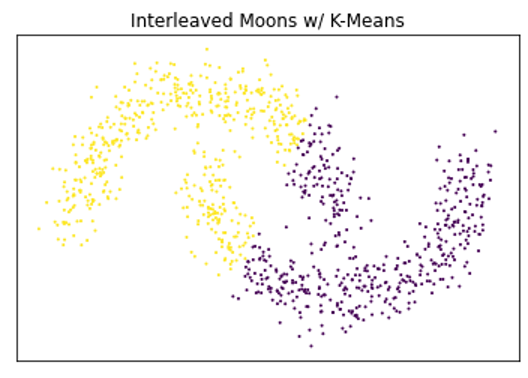
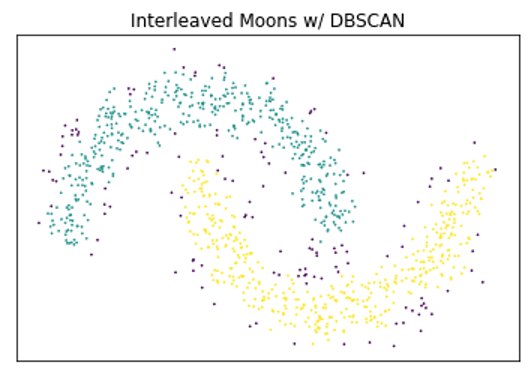
HDBSCAN 建立在一種眾所周知的基于密度的聚類算法 DBSCAN 的基礎上,該算法不要求提前知道簇的數量,但仍然存在一個不幸的缺點,即假設簇可以用一個全局密度閾值建模。這使得對具有不同密度的簇進行建模變得困難。 HDBSCAN 改進了這一缺點,通過使用單鏈聚簇來構建樹狀圖,從而可以找到不同密度的簇。另一種著名的基于密度的聚類方法稱為光學算法,它改進了 DBSCAN ,并使用分層聚類來發現密度不同的聚類。光學技術通過將點投影到一個新的空間(稱為可達空間)來改進標準的單鏈接聚類,該空間將噪聲從密集區域進一步移開,使其更易于處理。然而,與許多其他層次聚集聚類方法(如單鏈接聚類和完全鏈接聚類)一樣, OPTICS 也存在以單個全局切割值切割生成的樹狀圖的缺點。 HDBSCAN 本質上是光學+ DBSCAN ,引入了集群穩定性的度量,以在不同級別上切割樹狀圖。
我們將通過快速示例演示 HDBSCAN 的 RAPIDS cuML 實現中當前支持的功能,并將提供我們在 GPU 上實現的一些實際示例和基準。在閱讀了這篇博文之后,我們希望您對 RAPIDS ‘ GPU – 加速 HDBSCAN 實施可以為您的工作流和探索性數據分析過程帶來的好處感到興奮。
RAPIDS 中的 HDBSCAN 入門
GPU 提供了一組 RAPIDS – 加速 CPU 庫,幾乎可以替代 PyData 生態系統中許多流行的庫。下面的示例筆記本演示了 Python 上使用最廣泛的 HDBSCAN Python 庫與 GPU 上的 RAPIDS cuML HDBSCAN 之間的 API 兼容性(擾流板警報–在許多情況下,它與更改導入一樣簡單)。
下面是一個非常簡單的示例,演示了基于密度的聚類優于基于質心的技術對某些類型數據的好處,以及使用 HDBSCAN 優于 DBSCAN 的好處。
HDBSCAN 在實踐中的應用
基于密度的聚類技術自然適合于許多不同的聚類任務,因為它們能夠找到形狀奇特、大小各異的聚類。與許多其他通用機器學習算法一樣,沒有免費的午餐,因此盡管 HDBSCAN 改進了一些成熟的算法,但它仍然不是完成這項工作的最佳工具。盡管如此, DBSCAN 和 HDBSCAN 在從地理空間和協同過濾/推薦系統到金融和科學計算等領域的應用中取得了顯著的成功,被應用于從天文學到加速器物理學到基因組學等學科。它對噪聲的魯棒性也使得它對于異常值和異常檢測應用非常有用。
與數據分析和機器學習生態系統中的許多其他工具一樣,計算時間對生產系統和迭代工作流有很大的影響。更快的 HDBSCAN 意味著能夠嘗試更多的想法并制作更好的模型。下面是幾個使用 HDBSCAN 對單詞嵌入和單細胞 RNA 基因表達進行聚類的示例筆記本。這些都是為了簡短,并為您自己的數據集使用 HDBSCAN 提供了一個很好的起點。您是否已成功地將 HDBSCAN 應用于工業或科學領域,我們在此未列出?請留下評論,因為我們很想聽到。如果您在自己的硬件上運行示例筆記本電腦,還請告知我們您的設置以及您使用 RAPIDS 的經驗。
單詞嵌入
向量嵌入代表了一種流行且非常廣泛的聚類機器學習應用。我們之所以選擇 GoogleNews 數據集,是因為它足夠大,可以很好地顯示我們的算法的規模,但又足夠小,可以在一臺機器上執行。下面的筆記本演示了如何使用 HDBSCAN 查找有意義的主題,這些主題來自單詞嵌入的角度空間中的高密度區域,并使用 UMAP 可視化生成的主題簇。它使用整個數據集的一個子集進行可視化,但為調整不同的超參數和熟悉它們對結果集群的影響提供了一個很好的演示。我們使用默認的超參數設置(形狀為 3Mx300 )對整個數據集進行了基準測試,并在 24 小時后停止了 CPU 上的 Scikit learn contrib 實現。 RAPIDS 的實現大約需要 22 . 8 分鐘。
單細胞 RNA
下面是一個基于掃描和俱樂部庫中教程筆記本的工作流示例。本示例筆記本取自 RAPIDS 單單元示例存儲庫,其中還包含幾個筆記本,演示了 RAPIDS 用于單細胞和三級分析。在 DGX-1 (英特爾 40 核至強 CPU + NVIDIA V100 GPU )上,我們發現 HDBSCAN (在 GPU 上是~ 1s ,而不是具有多個 CPU 線程的~ 29s )使用了包含~ 70k 肺細胞基因表達的數據集上的前 50 個主成分,加速了 29x 。
在 GPU 上加速 HDBSCAN

RAPIDS CUML 項目包括端到端 GPU 加速的 HDBSCAN ,并提供 Python 和 C ++ + API 。與 cuML 中的許多基于鄰域的算法一樣,它利用 Facebook 的費斯庫中的蠻力 kNN 來加速相互可達空間中 kNN 圖的構建。這是目前的一個主要瓶頸,我們正在研究通過精確和近似近鄰選項進一步改進它的方法。
CUML 還包括單連鎖層次聚類的實現,它提供了 C ++和 Python API 。 GPU – 加速單個鏈接算法需要計算最小生成樹的新原語。此原語基于圖形,因此可以在 cugraph 和 cuml 庫中重用。我們的實現允許重新啟動,這樣我們就可以連接一個斷開連接的 knn 圖,并通過不必在 GPU 內存中存儲整個成對距離矩陣來提高可伸縮性。
與大多數CUML算法中的C++一樣,這些依賴于我們的大多數基于ML和基于圖元的[VZX27 ]。最后,他們利用利蘭·麥克因內斯和約翰·希利所做的偉大工作到 GPU ——甚至加快了群集壓縮和選擇步驟,使數據盡可能多地保留在 GPU 上,并在數據規模擴展到數百萬時提供額外的性能提升。
基準
我們使用了 McInnes 等人在 CPU 上的參考實現提供的基準筆記本,將其與 cuML 的新 GPU 實現進行比較。參考實現針對低維情況進行了高度優化,我們將高維情況與大量使用 Facebook FAISS 庫的暴力實現進行了比較。
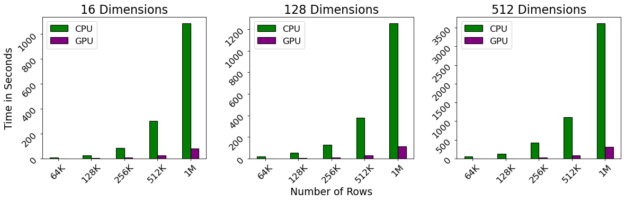
基準測試是在 DGX-1 上執行的,該 DGX-1 包含 40 核 Intel 至強 CPU 和 NVIDIA 32gb V100 GPU s 。即使對維度數進行線性縮放,對行數進行二次縮放,我們觀察到 GPU 仍然保持接近交互性能,即使行數超過 1M 。

發生了什么變化?

雖然我們已經成功地在 GPU 上實現了 HDBSCAN 算法的核心,但仍有機會進一步提高其性能,例如通過加快蠻力 kNN 圖構造刪除距離計算,甚至使用近似 kNN。雖然歐幾里德距離涵蓋了最廣泛的用途,但我們還想公開Scikit 學習 Contrib 實現中提供的其他距離度量。
scikit learn contrib 實現還包含許多不錯的附加功能,這些功能沒有包含在 HDBSCAN 上的開創性論文中,例如半監督和模糊聚類。我們也有堅固的單連桿和光學算法的構建塊,這將是 RAPIDS 未來的良好補充。最后,我們希望在將來支持稀疏輸入。
如果您發現這些功能中的一個或多個可以使您的應用程序或數據分析項目更成功,即使此處未列出這些功能,請轉到我們的Github 項目并創建一個問題。
概括
HDBSCAN 是一種相對較新的基于密度的聚類算法“站在巨人的肩膀上”,改進了著名的 DBSCAN 和光學算法。事實上,它的核心原語還增加了重用,并為其他算法提供了構建塊,例如基于圖的最小生成樹和 RAPIDS ML 和圖庫中的單鏈接聚類。
與其他數據建模算法一樣, HDBSCAN 并不是所有工作的完美工具,但它在工業和科學計算應用中都有很多實際用途。它還可以與 PCA 或 UMAP 等降維算法配合使用,尤其是在探索性數據分析應用中。
?

 by
by 







