細胞生物學和虛擬細胞模型的未來取決于大規模測量和分析數據。在過去 10 年里,單細胞實驗一直以驚人的速度增長,從數百個細胞開始,現在轉向使用數十億個細胞進行新的數據生成工作。
虛擬細胞模型還生成了數十億個虛擬細胞。大量數據和新開發的模型將幫助科學家發現新的生物學、開發新的療法,以及研究和闡明疾病和衰老的進展。
數據處理和分析是下游生物解釋和模型構建的關鍵。隨著數據的極端增長,出現了兩個關鍵的數據處理挑戰,極大地限制了對這些大規模數據集的科學認識和解釋:
- 數據規模:無法分析大型數據集 (millions to billions of cells)
- 分析速度:重要的專家級分析步驟需要數小時到數天的等待時間
RAPIDS 單細胞解決了單細胞數據處理、分析和集成的主要瓶頸
歸一化、降維、聚類和批量集成等分析步驟對于單細胞數據分析、解釋和模型開發至關重要。RAPIDS-singlecell 是一款經MIT許可的開源工具,由scverse開發,可應對數據規模和分析速度方面的挑戰。它通過CuPy和NVIDIA RAPIDS利用GPU加速,直接在社區標準AnnData數據結構上運行。
RAPIDS 單單元主要由 CuPy 庫提供支持,CuPy 庫幾乎可以直接替代 NumPy 和選定的 SciPy 函數,使用戶能夠編寫與標準 NumPy 語法高度一致的 Python 代碼,同時使用 NVIDIA GPU 的并行計算功能。使用的其他工具包括:
| RAPIDS 和 NVIDIA CUDA 庫 | 單細胞分析的示例任務 |
| NVIDIA cuML | 降維,包括 PCA、UMAP 和 t-SNE |
| NVIDIA cuGraph | 利用基于圖形的計算(包括 Leiden 和 Louvain)的單元聚類 |
| Dask | 通過跨多個 GPU 和節點的并行執行,擴展至超過 1 億個單元 |
| RAPIDS 內存管理器 | 數據自動溢出到主機內存,支持跨多個 GPU 架構進行大規模單細胞分析 |
| 降維,包括 PCA、UMAP 和 t-SNE | 使用 Python 編寫的即時編譯 CUDA 核函數,用于基因選擇/迭代圖形優化 |
利用單個節點上的數百萬個細胞擴展細胞科學的未來
在單細胞分析、真實數據和虛擬細胞實驗領域,數據規模挑戰是一個日益嚴峻的問題。隨著真實數據的增加和虛擬單元模型的開發,這個問題還在繼續增長。
AI 原生生物技術公司 Noetik 基于人類腫瘤和健康控制組織的 petabytes 空間數據開發了基礎模型 OCTO-vc。Noetik 正在使用包含 193M 個細胞的專有數據集構建多模態基礎模型,以模擬虛擬細胞和細胞系統。
“如果沒有加速計算,分析如此大規模的數據集是不可能的。借助 NVIDIA,我們的虛擬細胞實驗已經生成了超過 55 億個虛擬細胞。”Noetik 首席科學官 Jacob Rinaldi 說道。“我們現在不僅能夠支持這種規模的數據集,而且還能夠使用 NVIDIA RAPIDS 和 RAPIDS-singlecell 加速不同算法和數據集規模的分析。”
Rinaldi 的團隊利用 RAPIDS-singlecell,在 1.1M 個單元數據集上將 UMAP (12.85 分鐘到 1.64 秒) 和 Leiden 集群 (7.83 小時到 14.4 秒) 的速度分別提高了 470 倍和 1958 倍,將分析時間從使用 CPU 的數小時或數天縮短到了近乎實時。
由于表 1 中描述的效率,RAPIDS-singlecell 可以在幾秒鐘內容納數億個細胞。它還可以分析單個 GPU 上的數百萬個細胞。表 2 概述了使用 NVIDIA AI Blueprint 中提供的最新版 RAPIDS-singlecell 進行單細胞分析的最新基準測試。我們建議在使用 RAPIDS-singlecell 處理大型數據集時使用 Zarr 格式。
除非另有說明,否則這些基準測試來自 NVIDIA,并且位于單個 GPU 上。速度可能因數據集、GPU 實例和內存可用性而異。
| 針對 1M 個單元的單 GPU 基準測試 | ||||
| 工作負載 | 基準 | NVIDIA L40S GPU | NVIDIA RTX PRO 6000 服務器版 | NVIDIA DGX B200 |
| QC | 13.6 | 0.5 | 0.2 | 0.2 |
| 高度可變的基因 | 27.0 | 8.7 | 0.4 | 0.3 |
| 回歸 | 8.2 | 2.7 | 0.2 | 0.2 |
| 規模 | 15.4 | 0.3 | 0.2 | 0.1 |
| PCA | 141.0 | 18.1 | 2.0 | 1.2 |
| 所有預處理 | 313.0 | 40.0 | 4.1 | 2.9 |
| 近鄰 | 219.0 | 4.0 | 1.9 | 1.7 |
| UMAP | 574.0 | 2.4 | 1.7 | 1.2 |
| 魯萬聚類 | 422.0 | 4.4 | 1.8 | 1.5 |
| 萊頓聚類 | 1521.0 | 3.2 | 1.7 | 1.5 |
| tSNE | 2010.0 | 33.2 | 15.9 | 14.6 |
| 擴散貼圖 | 77.0 | 4.4 | 1.3 | 1.2 |
| 總處理時間 | 5176.0 | 92.0 | 28.4 | 24.6 |
managed_memory=True 。與運行 scanpy v1.11.1 的 CPU (AMD EPYC 7413 24-Core Processor 48 Threads) 相比利用 NVIDIA RAPIDS 和 NVIDIA Blackwell GPU 進行近乎實時的單細胞分析
借助 NVIDIA Blackwell GPU 的最新 RAPIDS-singlecell 支持,分析時間大幅縮短,逐漸接近單細胞數據的實時分析。
對于旨在探索細胞種群并深入研究亞集群或罕見細胞子集的科學家來說,這種工作流程至關重要。通過迭代運行降維和其他方法,他們可以從數據中發現新的生物學見解。
額外的 GPU 和新架構可縮短分析時間。PCA 在 Tahoe Bio 的 9500 萬個單元數據集上運行,具有 7000 個特征,在 Blackwell GPU 上可以在 10 秒內完成。表 3 顯示了 1100 萬個單元上的多 GPU 基準測試。
| 步驟 | NVIDIA RTX PRO 6000 服務器版 (8 個 GPU) | NVIDIA DGX B200 ( 8 個 GPU) |
| 對數歸一化 | 0.33 | 0.27 |
| 高度可變的基因 | 0.42 | 0.44 |
| 規模 | 0.59 | 0.53 |
| PCA | 1.62 | 1.73 |
| 近鄰 | 23.7 | 20.9 |
| UMAP | 10.5 | 11.7 |
| 萊頓聚類 | 18 | 17.6 |
使用 Harmony 引入加速的開源集成分析
特別是現在,當大型單細胞數據語料庫(包括 CZI cellxgene 和 Arc 的 Virtual Cell Atlas)的規模和復雜性不斷增加時,對工具的需求也在不斷增長,以幫助在各種實驗中集成數據集。對于分析和利用數據進行模型開發而言,這是一個非常有用的步驟。
RAPIDS-singlecell 更新了 Harmony 的優化實現,這是一種批量集成工具,可消除批量效應,幫助發現生物學見解。RAPIDS-singlecell 版本現已獲得 MIT 許可,并使用標簽向量編碼代替常用的 one-hot-encoding 矩陣。
在以下示例中,使用來自 CZI cellxgene 存儲庫的數據集,初始 UMAP 分析顯示,許多細胞按 assay 版本進行聚類。但是,在 Harmony 批量集成之后,其中許多批量效果被刪除,細胞類型開始出現。
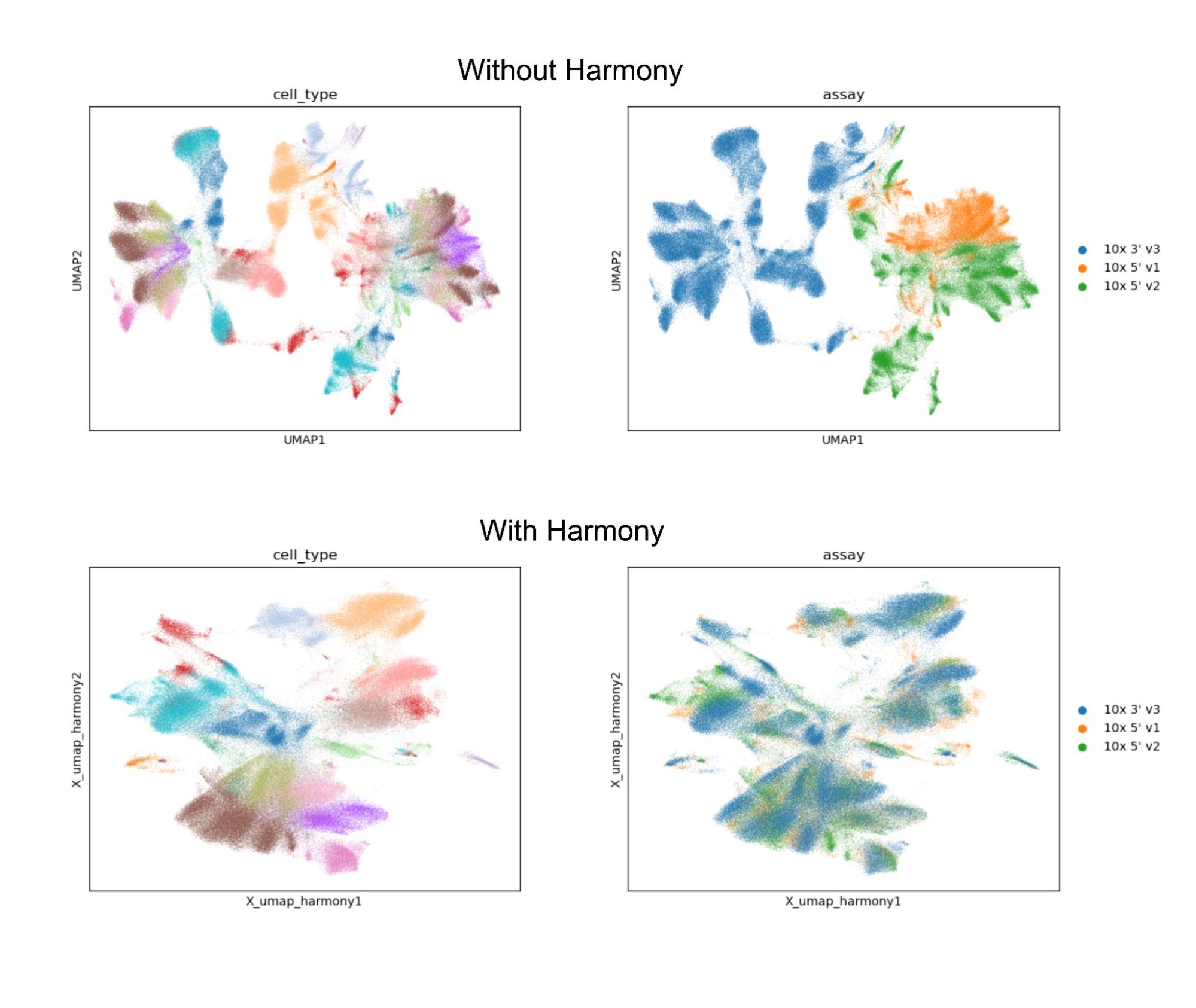
在處理 1100 萬個單元時,RAPIDS-singlecell上的 Harmony 可以比 CPU 快 350 倍以上,將分析時間從幾小時縮短到幾秒鐘,如表 4 所示。
| 出現次數 細胞 | 基準 | NVIDIA A10 Tensor Core GPU | NVIDIA L40S GPU | NVIDIA RTX PRO 6000 Server Edition | NVIDIA DGX B200 |
| 90000 | 120 | 3.3 | 2.6 | 1.6 | 1.6 |
| 200000 | 180 | 3.2 | 2.8 | 1.9 | 1.6 |
| 200 萬 | 1172 | 8 | 5.9 | 4.3 | 3.8 |
| 1100 萬 | > 7150 | 46.4 | 42.7 | 19.7 | 21.7 |
開始使用
以下實戰資源可幫助您開始使用 RAPIDS-singlecell。
- 來自 scverse 的 RAPIDS-singlecell 文檔
- 單細胞分析 Blueprint:一組可啟動的 Jupyter Notebook,引導用戶了解 RAPIDS-singlecell 的功能。可以手動部署,也可以通過 NVIDIA Brev 在預配置的云實例上部署。
- 加速數據科學和在數字生物學中利用基礎模型培訓課程展示了如何使用 RAPIDS-singlecell 清理數據集,以及如何使用數據重新訓練 Geneformer。它包含更多 Jupyter notebooks 以及隨附的幻燈片和錄制的演示文稿。
- NVIDIA 基因組學概述頁面涵蓋支持基因組學的 NVIDIA 工具。
致謝
我們要感謝 scverse 核心團隊,特別是 Philipp Angerer、Ilan Gold、Lukas Heumos 和 Issac Virshup,感謝他們提供的建議、見解和協作,以及 Corey Nolet 和 Avantika Lal 先前對 RAPIDS+single cell 數據進行的迭代和做出的貢獻。
還要感謝對單單元藍圖的重大貢獻,以及對 Harmony 的反饋 (按字母順序排列) :Alice Hsiung、Chelsea Gomatam、Daniel Burkhardt、Deven Yue、Eric Phan、Michelle Gill、Narges Masoudi 和 Seth Poulos,以及 Brev 團隊 (包括 Alec Fong、Anish Maddipoti、Carter Abdallah 和 Tyler Fong) 。
?