NVIDIA NGC 團隊正在舉辦一場網絡研討會,現場問答將深入了解 NGC 目錄中提供的這款 Jupyter 筆記本。學習如何使用這些資源來開啟你的人工智能之旅。立即注冊: NVIDIA NGC Jupyter 筆記本日:醫學影像分割 .
圖像分割通過將數字圖像的表示形式轉換為更有意義、更易于分析的內容,將數字圖像分割成多個部分。在醫學成像領域,圖像分割可以幫助識別器官和異常,測量它們,分類它們,甚至發現診斷信息。它通過使用從 x 射線、磁共振成像( MRI )、計算機斷層掃描( CT )、正電子發射斷層掃描( PET )和其他格式收集的數據來實現這一點。
為了實現能夠為用例提供所需精度和性能的最新模型,您必須設置正確的環境,使用理想的超參數進行訓練,并對其進行優化以達到所需的精度。所有這些都可能很耗時。數據科學家和開發人員需要一套合適的工具來快速克服繁瑣的任務。這就是我們建立 NGC 目錄的原因。
NGC 目錄 是 GPU 優化 AI 和 HPC 應用程序和工具的集線器。 NGC 提供了對性能優化容器的方便訪問,通過預訓練模型縮短了模型開發時間,并提供了特定于行業的 SDK 來幫助構建完整的 AI 解決方案和加快 AI 工作流。這些不同的資產可以用于各種用例,從計算機視覺和語音識別到語言理解。潛在的解決方案涵蓋汽車、醫療保健、制造和零售等行業。
![NGC catalog page shows cards for GPU-optimized HPC and AI containers, pretrained models, and industry SDKs that help accelerate workflows]](https://developer-blogs.nvidia.com/wp-content/uploads/2021/07/NGC-catalog-625x381.png)
基于 U-Net 的三維醫學圖像分割
在這篇文章中,我們將展示如何使用 醫學三維圖像分割筆記本 在 MRI 圖像中預測腦腫瘤。這個職位是適合任何人誰是新的人工智能和有一個特別的興趣在圖像分割,因為它適用于醫學成像。 3D-U-Net 實現了三維體的無縫分割,具有較高的精度和性能。它可以用來解決許多不同的分割問題。
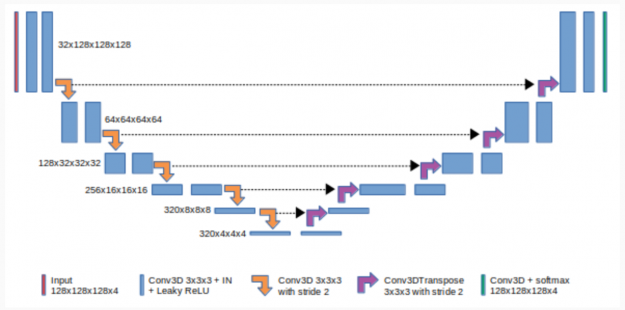
圖 2 顯示了 3du-Net 由收縮(左)和擴張(右)路徑組成。它重復應用未添加的卷積,然后使用最大池進行下采樣。
在深度學習中,卷積神經網絡( CNN )是深度神經網絡的一個子集,主要用于圖像識別和圖像處理。 CNN 使用深度學習來執行生成性和描述性任務,通常使用機器視覺以及推薦系統和自然語言處理。
CNNs 中的 Padding 指的是 CNN 內核處理圖像時添加到圖像中的像素數。未添加的 CNNs 意味著沒有像素添加到圖像中。
合用是 CNN 的一種下采樣方法。最大池是一種常見的池方法,它總結了功能最活躍的存在。擴展路徑中的每一步都包括特征映射的上采樣和與壓縮路徑中相應裁剪的特征映射的連接。
Requirements
此資源包含一個 Dockerfile ,它擴展了 TensorFlow NGC 容器并封裝了一些依賴項。可以使用以下命令下載資源:
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip
除了這些依賴項之外,還需要以下組件:
- NVIDIA 碼頭工人
- NGC 最新 TensorFlow 集裝箱
- NVIDIA 安培結構, NVIDIA 圖靈,或 NVIDIA 伏特 GPU
要使用具有張量核心的混合或 TF32 精度或使用 FP32 來訓練模型,請使用腦腫瘤分割數據集上 3D U-Net 模型的默認參數執行以下步驟。
下載資源
通過單擊 資源頁 右上角的三個點手動下載資源。
也可以使用以下 wget 命令:
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip
構建 U-Net TensorFlow NGC 容器
此命令使用 Dockerfile 創建一個名為 unet3d_tf 的 Docker 映像,自動下載所有必需的組件。
docker build -t unet3d_tf .
下載數據集
數據可在 腦腫瘤分割數據集 網站注冊獲得。應下載數據并將其放置在容器中安裝 /data 的位置。
運行容器
要在 NGC 容器中啟動交互式會話以運行預處理、訓練和推斷,必須運行以下命令。這將啟動容器并將 ./data 目錄作為卷裝載到容器中的 /data 目錄,將 ./results 目錄裝載到容器中的 /results 目錄。
使用容器的優點是它將所有必需的庫和依賴項打包到一個單獨的、隔離的環境中。這樣您就不必擔心復雜的安裝過程。
mkdir data
mkdir results
?
docker run --runtime=nvidia -it --shm-size=1g --ulimit memlock=-1 --ulimit stack=67108864 --rm --ipc=host -v ${PWD}/data:/data -v ${PWD}/results:/results? -p 8888:8888 unet3d_tf:latest /bin/bash
啟動容器內的筆記本
使用此命令在容器內啟動 Jupyter 筆記本:
jupyter notebook --ip 0.0.0.0 --port 8888 --allow-root
使用以下命令將數據集移動到容器內的/ data 目錄。 下載筆記本 :
?wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/med_3dunet/versions/1/zip -O med_3dunet_1.zip
然后,將下載的筆記本上傳到 JupyterLab 中,運行筆記本的單元格對數據集進行預處理,并對模型進行訓練、基準測試和測試。
Jupyter 筆記本
通過運行這個 Jupyter 筆記本的細胞,你可以首先檢查下載的數據集并看到腦腫瘤圖像。然后,查看數據預處理命令,準備數據進行訓練。下一步是訓練模型,并使用訓練過程中的檢查點作為預測步驟。最后,直觀地檢查預測函數的輸出。
檢查圖像
要檢查數據集,可以使用 nibabel ,這是一個提供對一些常見的醫學和神經成像文件格式的讀/寫訪問的包。
通過運行接下來的三個單元格,您可以使用 pip install 安裝 nibabel ,從數據集中選擇一個映像,并使用 matplotlib 從數據集中打印所選的第三個映像。您可以通過更改代碼中的圖像地址來檢查其他數據集圖像。
import nibabel as nib
import matplotlib.pyplot as plt
?
img_arr = nib.load('/data/MICCAI_BraTS_2019_Data_Training/HGG/BraTS19_2013_10_1/BraTS19_2013_10_1_flair.nii.gz').get_data()
?
def show_plane(ax, plane, cmap="gray", title=None):
??? ax.imshow(plane, cmap=cmap)
??? ax.axis("off")
?
??? if title:
??????? ax.set_title(title)
???????
(n_plane, n_row, n_col) = img_arr.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
?
show_plane(a, img_arr[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, img_arr[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, img_arr[:, :, n_col // 2], title=f'Column = {n_col // 2}')
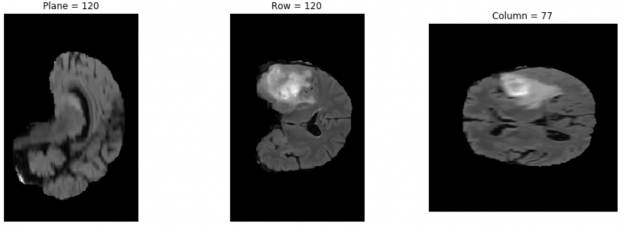
結果如圖 4 所示。
數據預處理
dataset/preprocess_data.py 腳本將原始數據轉換為用于培訓和評估的 TFRecord 格式。該數據集來自 2019 年布拉特挑戰賽 ,包含超過 3 TB 的多機構、常規、臨床獲得、術前、多模式、膠質母細胞瘤( GBM / HGG )和低級別膠質瘤( LGG )的 MRI 掃描,并經病理證實診斷。如果可用,還包括患者的總體生存率( OS )數據。這些數據是在訓練、驗證和測試數據集中構建的。
圖像的格式是 nii.gz. NIfTI 是神經成像的一種文件格式。可以通過運行以下命令對下載的數據集進行預處理:
python dataset/preprocess_data.py -i /data/MICCAI_BraTS_2019_Data_Training -o /data/preprocessed -v
處理后的圖像的最終格式是 tfrecord 。為了幫助您高效地讀取數據,請序列化數據并將其存儲在一組文件中(每個文件大約 100 到 200 MB ),每個文件都可以線性讀取。如果數據是通過網絡傳輸的,這一點尤其正確。它還可以用于緩存任何數據預處理。 TFRecord 格式是一種用于存儲二進制記錄序列的簡單格式,它大大加快了數據加載過程。
使用默認參數進行培訓
啟動 Docker 容器后,可以使用默認的超參數(例如,{ 1 到 8 } GPU s { TF-AMP / FP32 / TF32 })開始單個折疊(折疊 0 )的訓練:
Bash examples/unet3d_train_single{_TF-AMP}.sh <number/of/gpus> <path/to/dataset> <path/to/checkpoint> <batch/size>
例如,要以 32 位精度( FP32 或 TF32 )在一個 GPU 上以批大小 2 運行,請運行以下命令:
bash examples/unet3d_train_single.sh 1 /data/preprocessed /results 2
要訓練具有混合精度( TF-AMP )的單折疊,每個 GPU 有八個 GPU 個,每 GPU 批大小為 2 ,請運行以下命令:
bash examples/unet3d_train_single_TF-AMP.sh 8 /data/preprocessed /results 2
培訓績效基準
可以通過運行基準腳本來評估培訓績效:
bash examples/unet3d_{train,infer}_benchmark{_TF-AMP}.sh <number/of/gpus/for/training> <path/to/dataset> <path/to/checkpoint> <batch/size>
此腳本使模型運行并報告性能。例如,要在四個 GPU 上使用批量為 2 的 TF-AMP 對培訓進行基準測試,請運行以下命令:
bash examples/unet3d_train_benchmark_TF-AMP.sh 4 /data/preprocessed /results 2
Predict
您可以使用測試數據集和 predict as exec 模式來測試模型。結果保存在 model_dir 目錄中, data_dir 是數據集的路徑:
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test
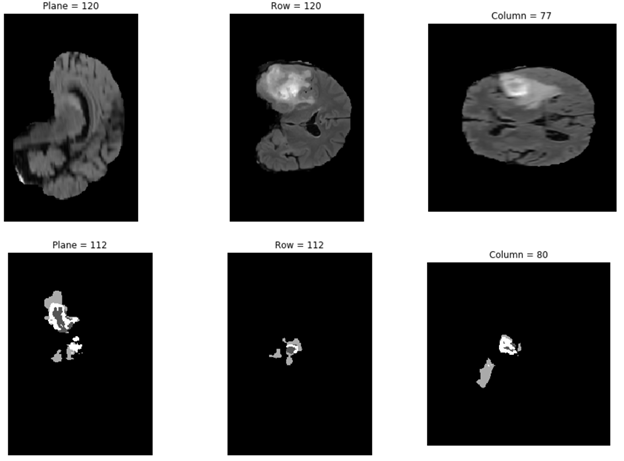
繪制預測結果
在下面的代碼示例中,您將從 results 文件夾打印所選結果之一:
import numpy as np
from mpl_toolkits import mplot3d
import matplotlib.pyplot as plt
data= np.load('/results/vol_0.npy')
def show_plane(ax, plane, cmap="gray", title=None):
??? ax.imshow(plane, cmap=cmap)
??? ax.axis("off")
??? if title:
??????? ax.set_title(title)???????
(n_plane, n_row, n_col) = data.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, data[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, data[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, data[:, :, n_col // 2], title=f'Column = {n_col // 2}')
高級選項
對于那些希望探索此筆記本內置高級功能的用戶,可以使用 -h 或 --help 查看 main . py 可用選項的完整列表。通過運行下一個單元格,您可以看到如何更改此腳本的執行模式和其他參數。使用此腳本,可以使用自定義的超參數執行模型訓練、預測、評估和推斷。
python main.py --help
main.py 參數可以更改以執行不同的任務,包括訓練、評估和預測。
也可以使用默認超參數訓練模型。通過運行 python main.py --help 命令,可以看到可以更改的參數列表,包括訓練超參數。例如,在訓練模式下,可以使用以下命令將學習速率從默認的 0 . 0002 更改為 0 . 001 ,并將訓練步驟從 16000 更改為 1000 :
python main.py --model_dir /results --exec_mode train --data_dir /data/preprocessed_test --learning_rate 0.001 --max_steps 1000
您可以運行 main.py 中提供的其他執行模式。例如,在本文中,我們通過運行以下命令使用了 python.py 的預測執行模式:
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test
總結和下一步
在這篇文章中,我們展示了如何使用一個簡單的 NGC 目錄中的 Jupyter 筆記本 開始使用醫學成像模型。當您從這個 Jupyter 筆記本轉換到構建您自己的醫學成像工作流時,考慮使用 NVIDIA Clara 列。 Clara 列車 包括人工智能輔助注釋 API 和注釋服務器,可以無縫地集成到任何醫療查看器中,使其具有 AI 能力。培訓框架包括分散式學習技術,例如針對 AI 工作流的聯合學習和轉移學習。
要了解如何使用這些資源并啟動您的人工智能之旅,請使用 live Q & A 注冊即將到來的網絡研討會, NVIDIA NGC Jupyter 筆記本日:醫學影像分割 。
?