創造新的候選藥物是一項英勇的努力,通常需要 10 多年的時間才能將一種藥物推向市場。理解生物學和化學文本的新的超級計算規模的大型語言模型( LLM )正在幫助科學家理解蛋白質、小分子、 DNA 和生物醫學文本。
這些最先進的人工智能模型有助于生成 de novo 蛋白質和分子,并預測蛋白質的 3D 結構。他們可以預測小分子與蛋白質的結合結構,并為科學家提供更容易的方法來設計新的候選藥物,最終為患者帶來希望。
2021 Exscientia brought an AI-designed drug candidate 進行臨床試驗后,其他幾家公司宣布他們的候選人正在試驗中。在專注于基于人工智能的發現的制藥公司中,有關于 160 discovery programs 的公開信息,據報道其中 15 種產品正在臨床開發中。
基于人工智能的藥物發現的前沿是 generating high-quality proteins 等應用的生成人工智能模型。這些大型、強大的模型從多 GPU 、多節點、高性能計算( HPC )基礎設施上的未標記數據(如測序數據)中學習。
有了 NVIDIA BioNeMo Service ,生物生成人工智能的工作流程得到了優化和交鑰匙。你可以專注于使人工智能模型適應合適的候選藥物,而不是處理配置文件和建立超級計算基礎設施。
生物 NeMo 服務
Bio NeMo 服務是早期藥物發現中生成人工智能的云服務,在一個地方擁有九個最先進的大型語言和擴散模型。
Bio NeMo 中的模型可以通過 web 界面或完全管理的 API 訪問,并且可以在 NVIDIA DGX Cloud 上進行進一步的訓練和優化。
使用 Bio NeMo 服務,您可以執行以下任何任務:
- 生成大型蛋白質庫。
- 使用嵌入來完善蛋白質庫,構建屬性預測因子。
- 生成具有特定財產的小分子。
- 快速準確地預測和可視化數十億蛋白質的 3D 結構。
- 開展大規模的配體到小分子姿態估計活動。
- 下載蛋白質、分子和預測的 3D 結構。
生物 NeMo 服務中的生成人工智能模型
Bio NeMo 服務有九個人工智能生成模型,涵蓋了開發人工智能藥物發現管道的廣泛應用:
- AlphaFold 2 、 ESMFold 和 OpenFold 用于從初級氨基酸序列預測 3D 蛋白質結構
- ESM-1nv 和 ESM-2 用于蛋白質性質預測
- ProtGPT2 用于蛋白質生成
- 用于小分子生成的 MegaMolBART 和 MoFlow
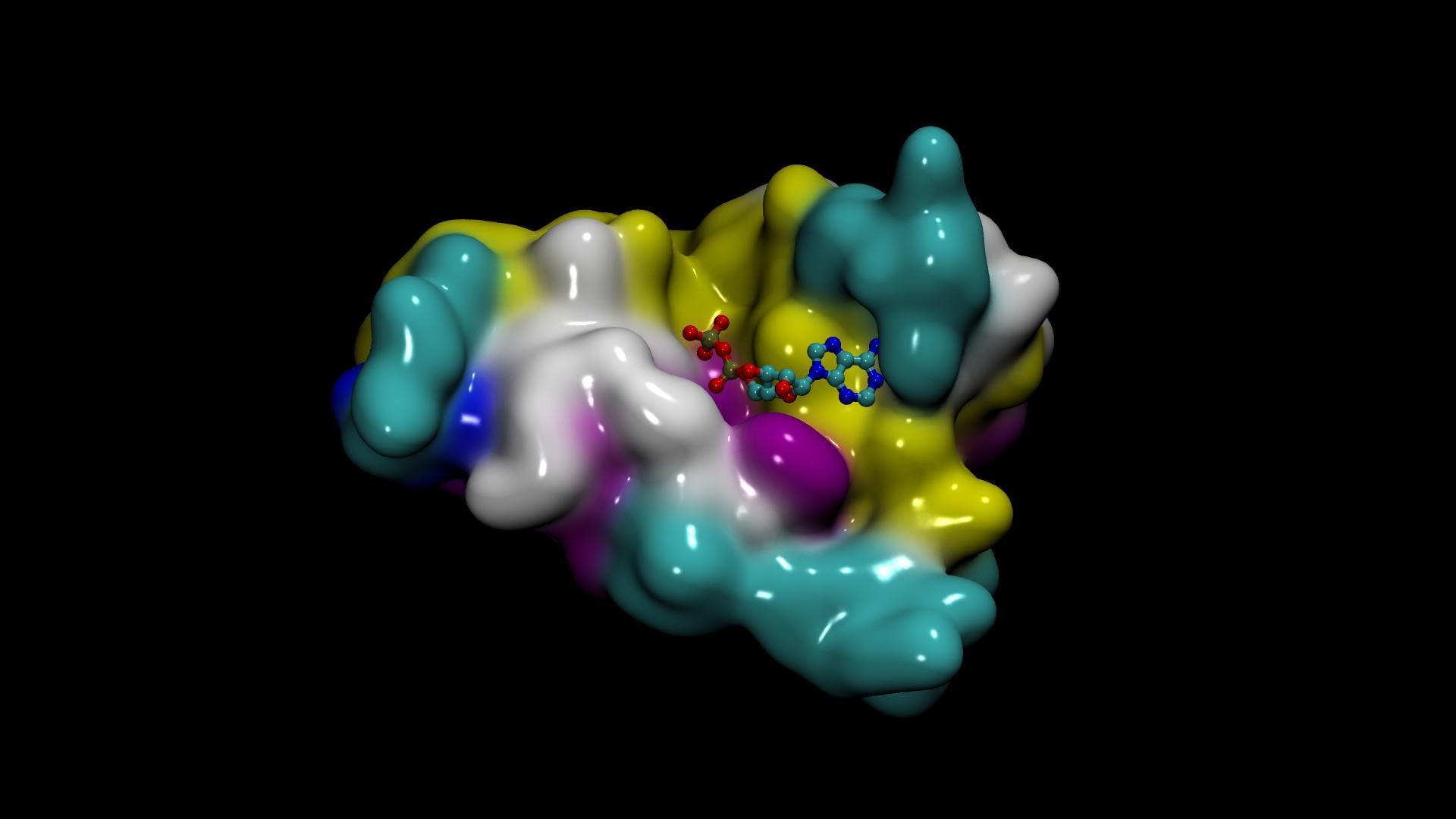
- DiffDock 用于預測小分子與蛋白質的結合結構
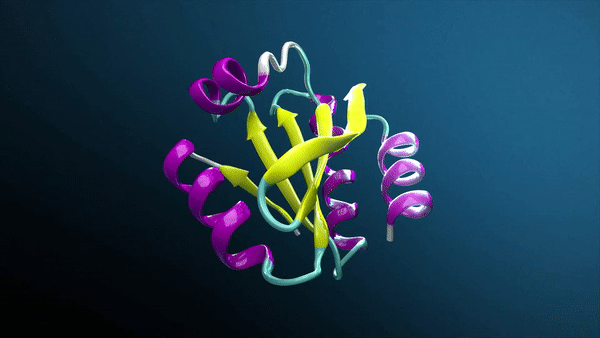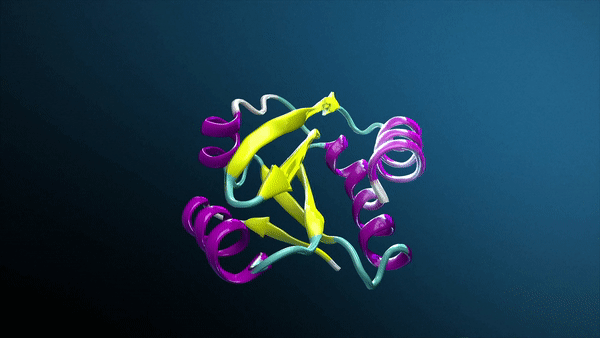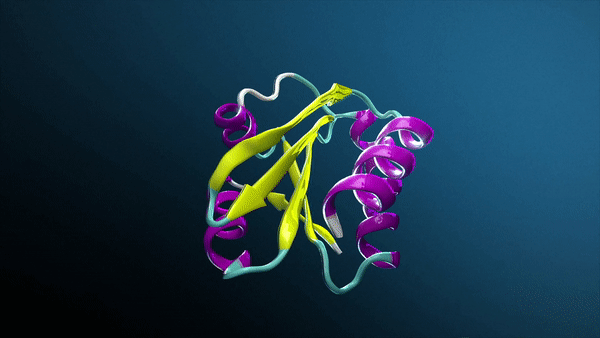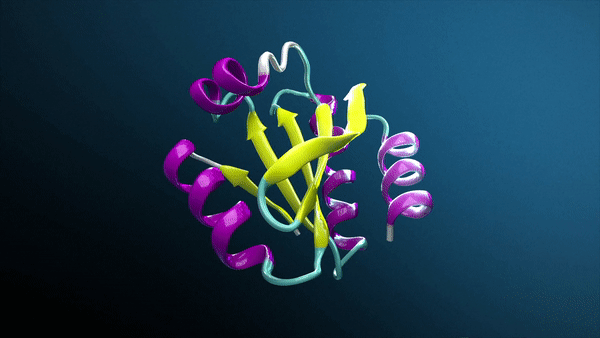
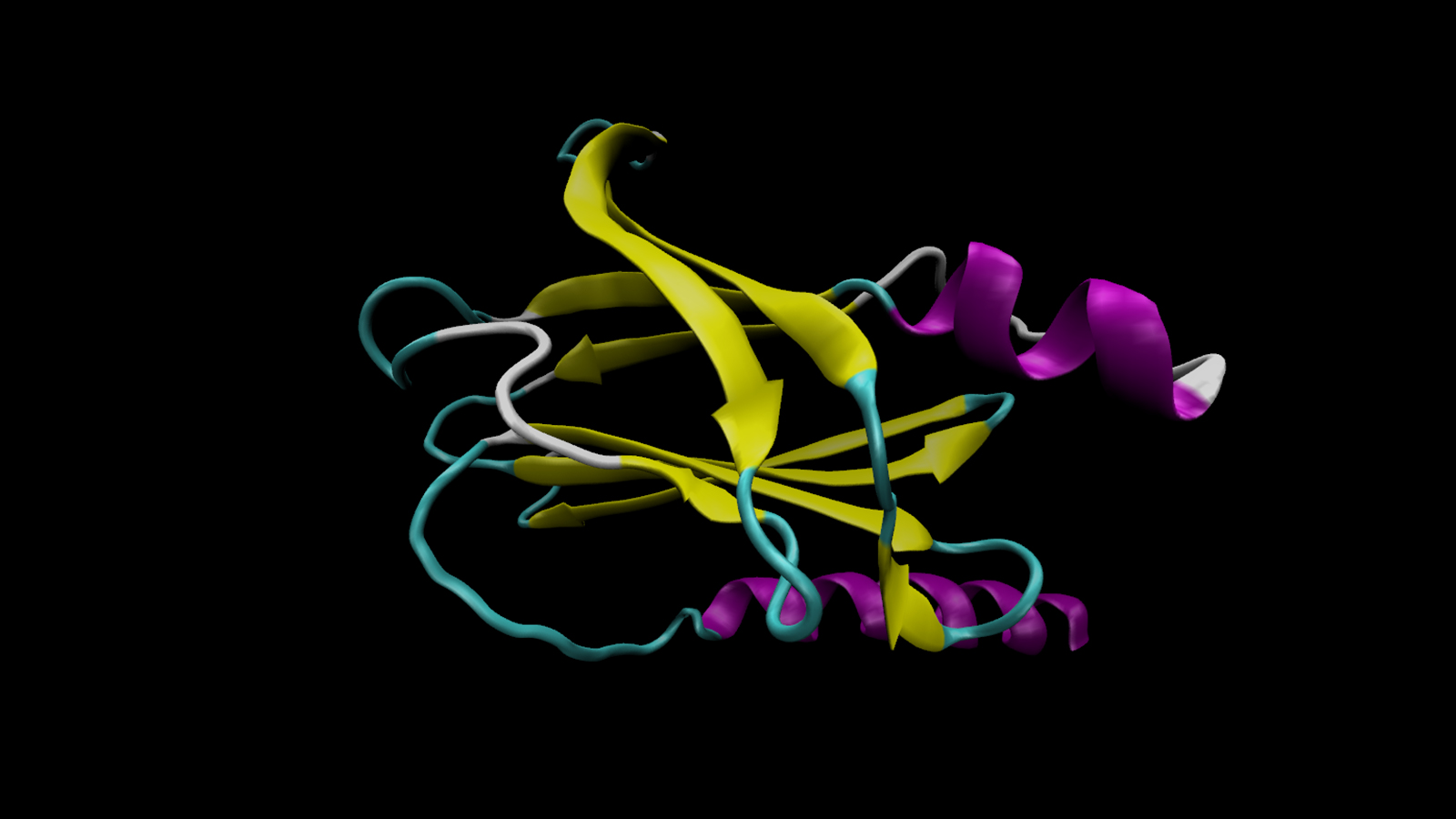
3D 蛋白質結構預測
蛋白質結構預測模型使科學家能夠從其初級線性氨基酸序列預測 3D 蛋白質結構。 AlphaFold 2 、 OpenFold 和 ESMFold 是 Bio NeMo 服務中可用于蛋白質結構預測的模型。
DeepMind 的 AlphaFold 2 在 CASP14, 達到了一個重要的里程碑,它在預測蛋白質 3D 結構方面達到了接近實驗的準確性。 AlphaFold 2 使用深度學習并在 JAX 中開發,即使只有少數同源序列可用,也能高精度預測蛋白質的氨基酸序列與其 3D 結構之間的關系。
OpenFold 是 DeepMind 的 AlphaFold 2 模型的忠實復制,用于從初級氨基酸序列預測 3D 蛋白質結構。雖然 AlphaFold 2 是在 JAX 工作流中開發的,但 OpenFold 的代碼基于 PyTorch 。 OpenFold 實現了與原始模型相似的精度,并以 0.96 ? RMSD95 的精度預測了中值主干。
Bio NeMo 將 OpenFold 加速了 6 倍,這樣藥物發現研究人員就可以分析更大的數據集并進行更多的迭代。 Bio NeMo 中的 OpenFold 也是可訓練的,這意味著可以為專門研究創建變體。
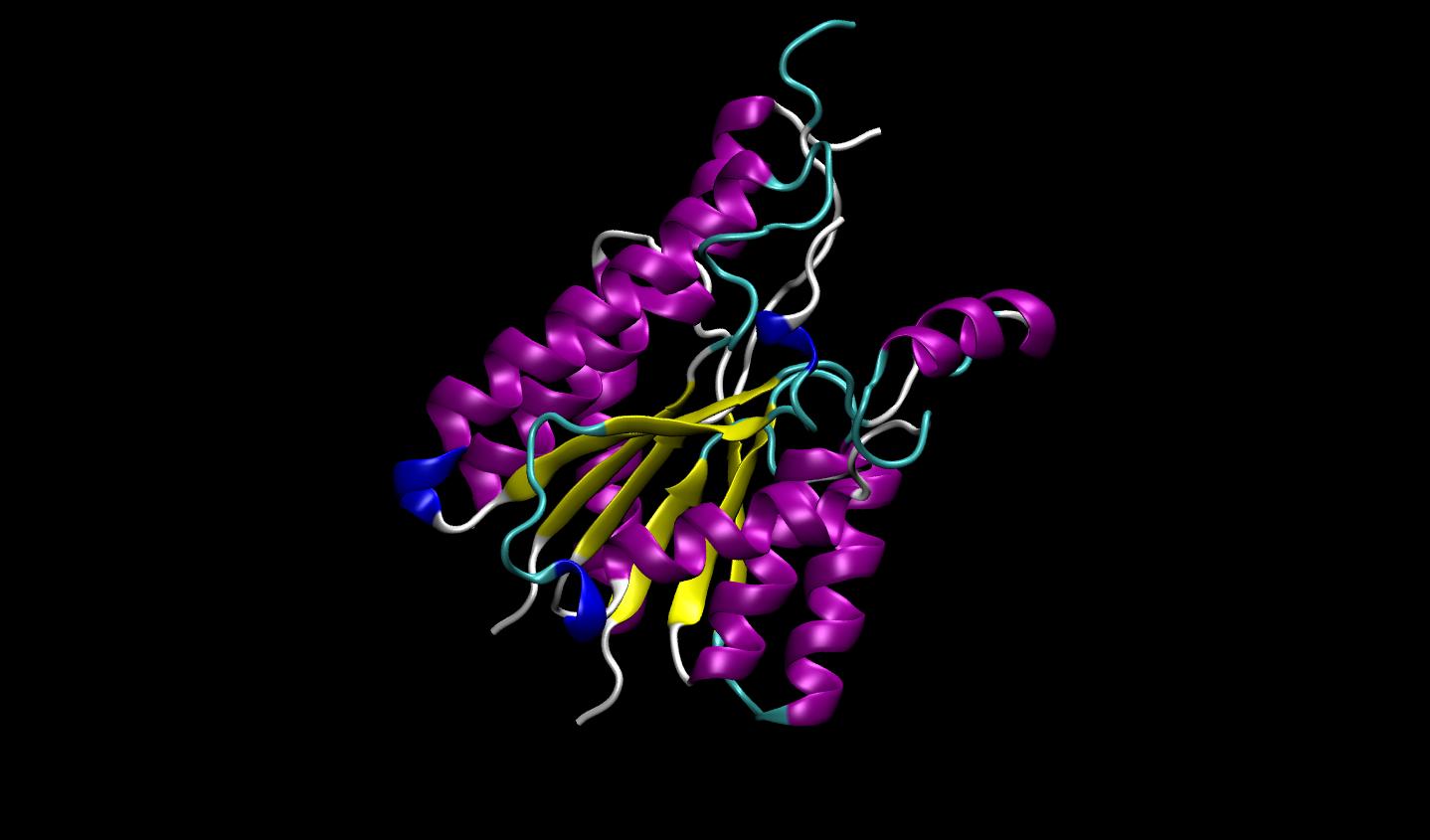
Meta 的 ESMFold 是一種基于 transformer 的、超快的 3D 蛋白質結構預測,基于 ESM-2 嵌入,無需多序列比對( MSA )。它包括一個折疊頭,可以實現完全端到端的單序列結構預測器。該模型通過無監督預訓練學習蛋白質序列,可以預測單個蛋白質序列的結構,而不需要許多同源序列作為輸入。
在單個 NVIDIA GPU 上, ESMFold 在 14.2 秒內預測出 384 個殘基的蛋白質,比單個 AlphaFold 2 模型快 6 倍。在較短的序列上,改進增加到~ 60 倍。
蛋白質性質預測
Meta 最先進的 ESM-1 and ESM-2 是用于蛋白質進化規模建模的 LLM 。他們受到 BERT 架構的啟發,并以掩蔽語言建模為目標,在數百萬個蛋白質序列上進行訓練。 ESM-1 和 ESM-2 了解了最終產生 2D 蛋白質結構和功能的氨基酸之間的模式和依賴性。
Bio NeMo 中的 ESM-1nv 是 Meta 的 ESM-1b 的忠實復制, ESM-1b 是蛋白質進化規模建模的 LLM 。它基于 BERT 架構,并在數百萬個蛋白質序列上進行訓練,目標是掩蔽語言建模。 ESM-1nv 學習最終產生蛋白質結構和功能的氨基酸之間的模式和依賴性。
通過 Bio NeMo , ESM-1nv 經過優化,可在大型計算基礎設施(如 DGX Cloud )上重新訓練、擴展和微調。
當 ESM-1nv 通常用于從氨基酸序列預測多種蛋白質財產時, ESM-2 通常用于預測突變對蛋白質穩定性的影響。
在 Bio NeMo 服務中嵌入 ESM-1nv 和 ESM-2 可以用于擬合下游任務模型,以了解感興趣的蛋白質財產,例如亞細胞位置、熱穩定性、水溶性和保守區域或可變區域。這是通過訓練一個通常小得多的模型來實現的,該模型具有監督學習目標,以從蛋白質序列的嵌入中推斷特性。這種方法已被證明在一系列預測任務上提供了最先進的準確性。
小分子生成
MegaMolBART 和 MoFlow 是生物 NeMo 中的生成化學 AI 模型。 MegaMolBart 是一個基于 transformer 的大型生成化學模型,用于分子優化。它使用 SMILES ,一種表示小分子化學結構的字符串表示法。由阿斯利康和 NVIDIA 開發的 MegaMolBART 最適合生成具有實驗測試的結合親和力的新小分子,以及用于分子嵌入。
MegaMolBART 依賴于 NVIDIA NeMo ,它為開發、訓練和部署深度學習模型(包括 Megatron 模型)提供了一個強大的環境。
Megatron 為 PyTorch 照明提供了增強功能,例如具有 YAML 文件和檢查點管理的超參數可配置性。它還允許使用 NVIDIA NeMo – NeMo 開發和訓練大型 transformer 模型,這使得具有數據并行性、模型并行性和混合精度的多 GPU 、多節點訓練易于配置。
使用 ZINC-15 數據庫對 MegaMolBART 進行預訓練。從滿足以下限制的部分中選擇了大約 14.5 億個分子( SMILES 串):
- 分子量<= 500 道爾頓
- 對數 P <= 5
- 反應性水平為“反應性”
- 可購買性被“注釋”
來自威爾康奈爾醫學院團隊的基于流的生成模型 MoFlow 學習分子圖及其潛在表示之間的可逆映射。由深度圖生成模型驅動生成具有所需化學財產的分子圖可以加速藥物發現過程。
MoFlow 通過一種新穎的、基于條件流的方法實現了最先進的性能。它使用圖卷積,并產生一個遵循鍵價約束的有效分子圖。它用于分子生成、重建和優化。
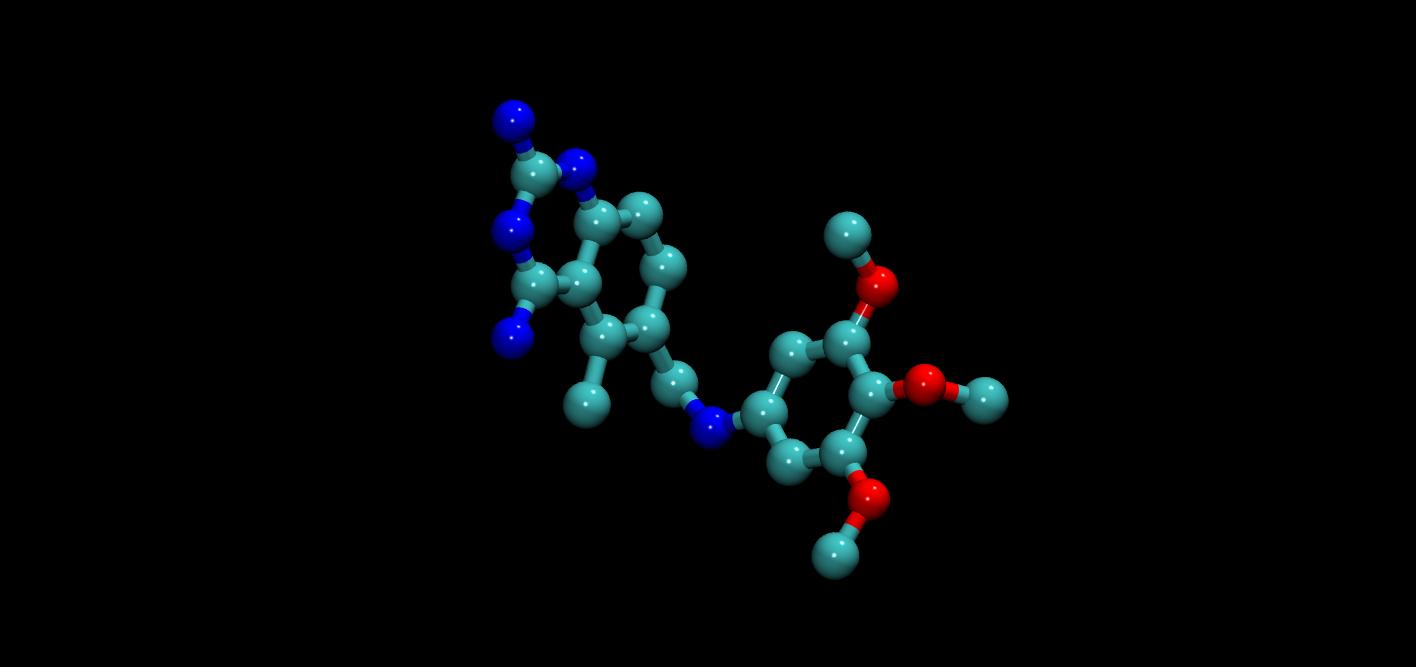
蛋白質生成
ProtGPT2 由 ISMB 和德國拜勒大學創建,是一種基于 GPT2 transformer 體系結構的 LLM ,可生成 de novo 蛋白質序列,以識別獨特的結構、財產和功能。當訓練數據有限時,該模型對于生成自定義蛋白質序列是最優的。
它是在蛋白質空間數據庫 UniRef50 上訓練的,有 36 層,參數為 738M 。使用因果建模目標來訓練 ProtGPT2 ,其中訓練模型以預測序列中的下一個標記(或在這種情況下,低聚物)。通過這樣做,該模型學習了蛋白質的內部表示,并可以說蛋白質語言。
分子對接
麻省理工學院 Jameel 診所的 DiffDock 是一個擴散生成人工智能模型。它預測小分子配體與蛋白質的結合結構,稱為 molecular docking 或 pose prediction 。
DiffDock 具有快速的推理時間,并提供具有高選擇性精度的置信度估計。該模型具有高度的準確性和計算效率。它在 PDBBind 盲對接基準上實現了新的最先進的 38% 的前 1 預測, RMSD < 2A ,大大超過了以前的最佳搜索( 23% )和深度學習方法( 20% )。
DiffDock 在 PDBBind 基準的分子復合物上進行了評估,并與最先進的基于搜索的方法(如 SMINA 和 GLIDE )以及最近的深度學習方法 EquiBind 和 TANKBind 進行了比較。 DiffDock 可以幫助人工智能藥物發現管道,并為下游任務集成開辟新的研究途徑。
開始使用 Bio NeMo
要了解更多關于 Bio NeMo 的信息并申請盡早訪問 Bio NeMo 服務,請參閱 BioNeMo 頁面。
本周, NVIDIA GTC 2023 在 latest AI advances in drug discovery 上舉行了多次會議。免費注冊,即可按需訪問所有內容,并查看以下關于人工智能藥物發現和生物 NeMo 的課程:
- A Transformative A I Platform to Accelerate Biologics Discovery
- Generative Deep Learning with BioNeMo for Protein Therapeutics
- AI-Powered Drug Discovery
- Understanding the Chemical and Biological Language of Life with LLMs using BioNeMo
- Using AI to Accelerate Scientific Discovery
- Artificial Intelligence Captures the Language of Life Written in Proteins
?