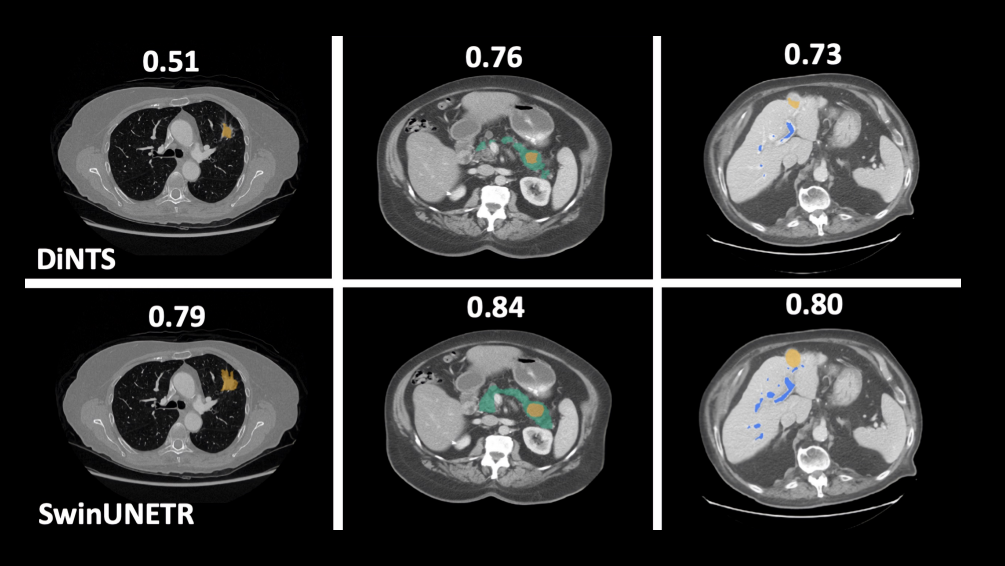
加州大學洛杉磯分校(UCLA)的研究人員開發了一種新的 AI 模型,該模型可以專業地分析疾病的 3D 醫學圖像,而所需的時間遠低于人類臨床專家所需的時間。
名為 SL I Vi T (SLice Integration by Vision Transformer) 的深度學習框架可分析來自不同圖像模式(包括視網膜掃描、超聲波視頻、CT、MRI 等)的圖像,識別潛在的疾病風險生物標志物。
領導這項研究的計算醫學專家兼加州大學洛杉磯分校(UCLA)教授埃蘭·哈爾珀林(Eran Halperin)博士表示,該模型在各種疾病中的準確性很高,優于許多現有的特定疾病基礎模型。該模型采用了一種新的預訓練和微調方法,依賴于可訪問的大型公共數據集。因此,哈爾珀林博士認為可以相對較低的成本部署該模型來識別不同的疾病生物標志物,從而實現專家級醫學成像分析的大眾化。
研究人員使用 NVIDIA T4 GPUs 和 NVIDIA V100 Tensor Core GPUs ,以及 NVIDIA CUDA ,進行了研究。
目前,醫學影像專家往往不堪重負。患者通常要等待數周才能獲得 X 光、MRI 或 CT 掃描的評估結果,然后才能開始治療。
SLIViT 的一個潛在優勢在于,它能夠以專業方式大規模分析患者數據,其專業知識也可以得到升級。例如,在開發新的醫學成像技術后,可以使用新數據對模型進行微調,這些新數據可以被推出并用于未來的分析。
Halperin 指出,該模型也易于部署。特別是在醫學影像專家稀缺的地方,未來該模型可能會對患者的治療效果產生重大影響。
Halperin 博士表示,在 SLIViT 問世之前,以人類臨床專家的級別評估大量掃描實際上是不可行的。借助 SLIViT,大規模、準確的分析成為現實。
Halperin 說:“該模型可以在識別疾病生物標志物方面產生顯著的影響,而無需使用大量手動標注的圖像。這些疾病生物標志物可以幫助我們了解患者的疾病軌跡。在未來,我們可以利用這些見解,根據通過 SLIVIT 發現的生物標志物為患者定制治療方案,并希望對患者的生活產生顯著的改善。”
根據加州大學洛杉磯分校(UCLA)研究人員在 《自然生物醫學工程》(Nature Biomedical Engineering)期刊上發表的一篇論文的主要作者 Oren Avram 博士指出,這項研究揭示了兩個驚人但相關的結果

首先,雖然該模型主要基于 2D 掃描數據集進行預訓練,但它能準確識別人體器官 3D 掃描中的疾病生物標志。通常,用于分析 3D 圖像的模型是在 3D 數據集上訓練的。但是,3D 醫療數據的獲取成本要高得多,因此比 2D 醫療數據要少得多且難以獲取。
加州大學洛杉磯分校 (UCLA) 的研究人員發現,通過在 2D 掃描圖像(更容易獲得)上預訓練模型,并在相對少量的 3D 掃描圖像上對其進行微調,該模型的表現優于僅在 3D 掃描圖像上訓練的專業模型。
第二個意想不到的結果是,該模型在遷移學習方面表現得如何。它通過微調由來自不同模式和器官的圖像組成的數據集,學會識別不同的疾病生物標志物。
Avram 說:“我們使用 2D 視網膜掃描——即您眼睛的圖像——對模型進行訓練,但隨后使用肝臟的 MRI 對模型進行了微調,這似乎沒有聯系,因為它們是兩個完全不同的器官和成像技術。”但我們了解到,視網膜和肝臟之間以及 OCT 和 MRI 之間存在一些共享的基本特征,這些特征可用于幫助模型進行下游學習,即使圖像領域完全不同。”
閱讀其他內容 新聞 來自加州大學洛杉磯分校關于 SLIViT 的文章。
請查看 SLIViT 論文: 通過使用 2D 掃描預訓練的深度視覺模型,從體積醫學掃描中準確預測疾病風險因素 。
在? GitHub 上訪問模型。
?